Die Core Unit CUMI gehört zu einer von insgesamt drei Core Units der Veterinärmedizinischen Fakultät und wird von wissenschaftlichen Mitarbeitern des Instituts für Bakteriologie und Mykologie geleitet. Wir verfügen über ein MALDI-TOF-Massenspektrometer, das die Identifizierung und Differenzierung von Mikroorganismen ermöglicht. Unser Institut besitzt mehrjährige Expertise in der Differenzierung von Bakterien und Pilzen über MALDI-TOF-MS.
Core Unit "Massenspektrometrische Identifizierung und Differenzierung von Mikroorganismen” (CUMI)
MALDI-TOF ist eine Massenanalyse, welche die Matrix–Assistierte Laser–Desorption–Ionisierung (MALDI) mit der Flugzeitanalyse (englisch: time of flight, TOF) verbindet. Da die MALDI-TOF-MS eine markierungsfreie Methode darstellt, die ohne Farbstoffe, Antikörper oder radioaktive Elemente auskommt, steht sie einem breiten Anwendungsspektrum zur Verfügung. Am Institut für Bakteriologie und Mykologie wird derzeit das microflex LT/microflex LRF MALDI-TOF-MS (Bruker Daltonics GmbH) genutzt, das die Identifizierung und Differenzierung von Mikroorganismen (Bakterien, Pilze und Hefen) ermöglicht. Darüber hinaus können Subtypisierung und gezielte Resistenzbestimmung innerhalb der methodischen Grenzen vorgenommen werden. In den nächsten Monaten wird das bisherige Modell durch ein leistungsfähigeres Gerät ersetzt, wodurch die Errichtung der neuen Core Unit "Massenspektrometrische Identifizierung und Differenzierung von Mikroorganismen” (CUMI) realisiert werden wird.
Die Core Unit wird von Prof. Wieland Schrödl und technischem Personal betreut. Bei der Planung und Durchführung anstehender Projekte stehen sie mit ihrer MALDI-TOF-MS-Expertise beratend zur Seite.
- Die Probe wird in eine niedermolekulare Matrix gegeben und auf einem Träger fixiert.
- Die Matrix-Proben-Mischung wird im Hochvakuum durch einen gepulsten fokussiertern Laserstrahl explosionsartig verdampft (Desorption).
- Die zu untersuchenden Analytmoleküle (Proteine) der Erreger werden ionisiert (Ionisierung).
- Anschließend werden die Ionen in einem starken elektrischen Feld beschleunigt und ihre jeweilige Flugzeit (TOF) registriert, wobei kleinere, leichtere
Ionen schneller am Detektor auftreffen. - Da die Flugzeit der Analyten von ihrer Masse (m) und ihrer Ladungszahl (z) abhängen, können die unterschiedlichen Massen der einzelnen Analyten bestimmt und als Gesamtspektrum dargestellt werden.
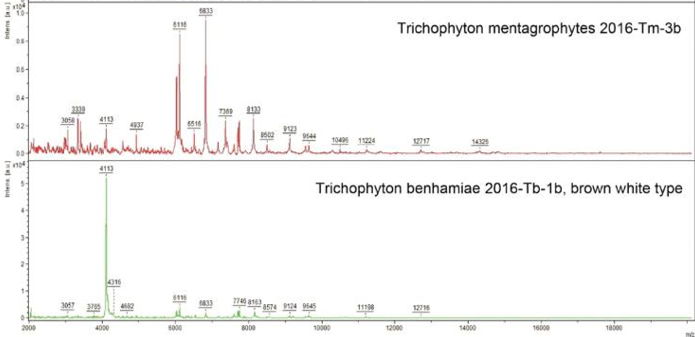
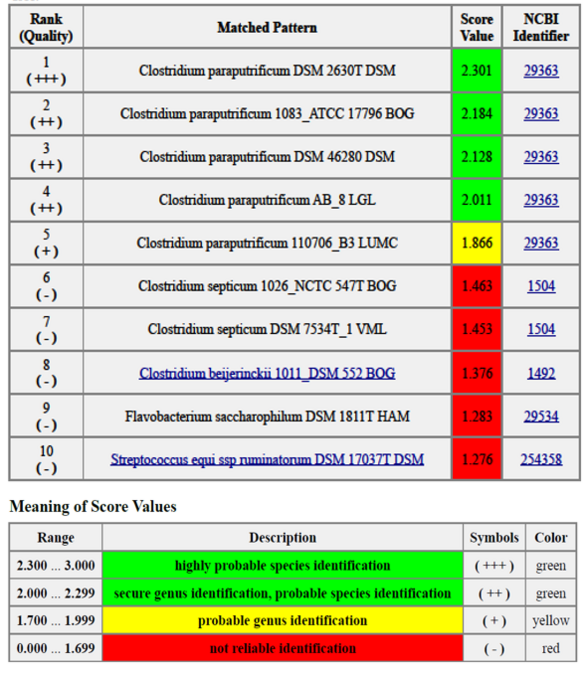
- Die jeweiligen Spektren sind für die einzelnen Bakterien und Pilze charakteristisch, wodurch eine Keimidentifizierung möglich ist. Dies erfolgt über den Abgleich der ermittelten Spektren mit einer im System integrierten Referenzdatenbank, die Spektren einer großen Zahl an humanmedizinisch und veterinärmedizinisch crelevanten Bakterien und Pilzen vergleicht.
- Beim Datenbank-Abgleich wird außerdem ein Zahlenwert (score) erzeugt, der Aufschluss über die Validität des Differenzierungsergebnisses gibt.
Da der Software-basierte Abgleich automatisiert und ohne Zeitverlust verläuft, erfolgt die Keimidentifizierung vom Auftragen einer Probe auf die Trägerplatte bis zum Vorliegen eines Differenzierungsergebnisses innerhalb weniger Minuten.
Die Spektren-Datenbank des Herstellers (Bruker Daltonik GmbH) wird jährlich aktualisiert und enthält derzeit 2462 Spezieseinträge, die einer Summe von 6904 main spectra/ Masterspektren (MSP) entsprechen. In den vergangenen 10 Jahren wurde diese Datenbank durch wissenschaftliche Mitarbeiter unseres Instituts durch das Einpflegen von über 3200 Masterspektren erheblich erweitert. Die Datenbank des Herstellers wurde insbesondere für folgende Erregergruppen ergänzt: Dermatophyten, Mucorales, Clostridium spec., Brachyspira spec., Rodentibacter pneumotropicus, Rodentibacter heylii und Muribacter muris. Die Datenbankerweiterungen zu den murinen Erregern wurden in Kooperation mit Bruker Daltonik GmbH durchgeführt. Weiterhin wurde die Datenbank über einen Datentransfer mit dem Robert-Koch Institut um die Gattungen Yersinia, Bacillus und Burkholderia erweitert.
Bei den Messungen durch interne und externe Nutzer werden die institutseigenen Datenbankerweiterungen berücksichtigt.
Die Core Unit steht unter Zugrundelegung einer aktuellen Gebührenordnung internen und externen Nutzern zur Verfügung. Dies betrifft Institute bzw. wissenschaftliche Mitarbeiter der Veterinärmedizinischen Fakultät als auch anderer Fakultäten der Universität Leipzig, externe wissenschaftliche Forschungseinrichtungen und privatwirtschaftliche Auftraggeber.
Die CUMI wird grundsätzlich als Operator-Einheit geführt, in der die Messungen von einer eingearbeiteten Person des Instituts durchgeführt werden. Dies betrifft insbesondere die routinemäßige Identifizierung von Mikroorganismen. Für die Bearbeitung besonderer Fragestellungen und umfangreicherer Projekte sind mit dem Institutsleiter bzw. dem Geräteverantwortlichen im Vorfeld spezifische Vereinbarungen zu treffen.
Weitere Informationen zur Nutzung der CUMI sowie die aktuellen Gebührensätze entnehmen Sie bitte der Nutzungsordnung
.
Für die Nutzung des MALDI-TOF-MS der CUMI ist das Ausfüllen des Auftragsformulars notwendig.
Die Anzucht der Kulturen und die Aufarbeitung der Proben erfolgt in der Regel durch den Nutzer. Für die MALDI-TOF-MS-Analyse wird eine kleine Menge einer Kolonie auf eine Analysenposition (Targetspot) des MALDI-Probenträgers aufgebracht. Auf die Proben wird anschließend ca. ein Mikroliter einer Matrix-Lösung (HCCA: α-Cyano-4-hydroxyzimtsäure) pipettiert. Der Probenträger wird mit dem ausgefüllten Auftragsformular, auf dem die Beschickung des Probenträgers einzutragen ist, zur Messung übergeben.
Grundsätzlich gewährleistet das Institut für Bakteriologie und Mykologie als Betreiber der CUMI, dass ein zur Messung erhaltener Probenträger an einem Arbeitstag innerhalb von 24 Stunden gemessen wird. Sollte dies ausnahmsweise nicht möglich sein, wird der Nutzer innerhalb dieser Zeit über die Verzögerung informiert.
Die Resultate (Identifizierungen) werden dem Nutzer in einem sogenannten Score-Report als PDF-Datei zur Verfügung gestellt. Hilfestellung und Erläuterungen werden dem Nutzer angeboten. Weitergehende Datenauswertungen in Zusammenarbeit mit dem Nutzer bedürfen dabei der gegenseitigen Absprache.
Sollte Ihnen die Anzucht der Kulturen und die Aufarbeitung der Proben nicht möglich sein, kontaktieren Sie bitte Prof. Wieland Schrödl, der mit Ihnen die weitere Vorgehensweise und die individuelle Preisgestaltung bespricht.
Die aktuellen Gebührensätz für die Keimidentifizierung und die Messung von Targetspots mittels der MALDI-TOF-MS-Technologie entnehmen Sie bitte der Nutzungsordnung
.
Die CUMI bietet die Möglichkeit mit Erregern der Risikogruppe 2 gemäß Biostoffverordnung (BioStoffV) zu arbeiten.